Taxonomie & séléction/3 : La structure génétique de la marijuana et du chanvre ( Indica /Sativa / Ruderalis )
La structure génétique de la marijuana et du chanvre
Abstrait
Malgré sa culture en tant que source de nourriture, de fibres et de médicaments, et son statut mondial en tant que drogue illicite la plus utilisée, le genre Cannabis a une organisation taxinomique peu concluante et une histoire évolutive. Les types de drogues de cannabis (marijuana), qui contiennent des quantités élevées de cannabinoïde psychoactif Δ 9 -tetrahydrocannabinol (THC), sont utilisés à des fins médicales et comme un médicament récréatif. Les types de chanvre sont cultivés pour la production de graines et de fibres et contiennent de faibles quantités de THC. Deux espèces ou des pools de gènes ( C . Sativa et C . Indica sont largement utilisés) pour décrire le pedigree ou l' apparence de cultiver le cannabisles plantes. En utilisant 14 031 polymorphismes mononucléotidiques (SNP) génotypés dans 81 échantillons de marijuana et 43 chanvre, nous montrons que la marijuana et le chanvre sont significativement différenciés au niveau du génome, démontrant que la distinction entre ces populations ne se limite pas aux gènes sous-jacents. Nous trouvons une corrélation modérée entre la structure génétique des souches de marijuana et leur C signalé . sativa et C . l' ascendance indica et montrent que les noms de souches de marijuana ne reflètent souvent pas une identité génétique significative. Nous fournissons également des preuves que le chanvre est génétiquement plus semblable à C . indica marijuana type qu'à C . sativa souches.
introduction
Le cannabis est l'une des cultures les plus anciennes de l'humanité, avec des antécédents d'utilisation datant de 6000 ans avant le présent. Peut-être en raison de ses origines précoces, et en raison des restrictions sur la recherche scientifique provoquée par la politique de drogue, l'histoire de l'évolution et de la domestication du cannabis reste mal comprise. Hillig (2005) proposée sur la base de allozyme variation que le genre se compose de trois espèces ( C . Sativa , C . Indica , et C . Ruderalis ) [ 1 ], alors qu'un autre point de vue est que le cannabis est monotypic et que les sous - populations observables représentent sous - espèce de C.sativa [ 2 ]. L'espèce putative C . ruderalis peut représenter des populations sauvages des autres types ou celles adaptées aux régions du nord.
La classification des populations de cannabis est confondue par de nombreux facteurs culturels, et retracer l'histoire d'une plante qui a connu une vaste dispersion géographique et une sélection artificielle par les humains pendant des milliers d'années s'est révélée difficile. Beaucoup de types de chanvre ont des noms variétaux tandis que les types de marijuana n'ont pas de système d'enregistrement horticole organisé et sont appelés souches. Le projet de génome et du transcriptome de C . sativa ont été publiés en 2011 [ 3 ], mais jusqu'à présent il n'y a pas eu d'étude publiée sur la structure de la population de cannabis utilisant des méthodes de génotypage à haut débit. Alors que l'opinion publique et la législation de nombreux pays se tournent vers la reconnaissance du cannabisEn tant que plante à valeur médicale et agricole [ 4 ], la caractérisation génétique de la marijuana et du chanvre devient de plus en plus importante pour la recherche clinique et les efforts d'amélioration des cultures.
Un premier pas important vers des analyses évolutives et fonctionnelles plus approfondies du cannabis , y compris la cartographie des caractères et l'identification de la variation génétique fonctionnelle, est la caractérisation de la structure génétique du genre. Ici, nous rapportons le génotypage d'une collection diversifiée de germoplasme de cannabis et montrons que les différences génétiques entre le chanvre et la marijuana ne sont pas limitées aux gènes impliqués dans la production de THC, alors que le C signalé . sativa et C . L' ascendance indica des variétés de marijuana ne capture que partiellement les principaux axes de la structure génétique de la marijuana.
Résultats et discussion
Pour évaluer la structure génétique du Cannabis communément cultivé , nous avons génotypé 81 échantillons de marijuana et 43 échantillons de chanvre en utilisant le génotypage par séquençage (GBS) [ 5 ]. Les échantillons de marijuana représentent un large éventail de souches commerciales et de variétés locales modernes, tandis que les échantillons de chanvre comprennent diverses accessions européennes et asiatiques et des variétés modernes. Bien que nous ayons échantillonné une variété de types de cannabis dans notre étude, l'accès aux échantillons est compliqué par le fait que la marijuana est une drogue illicite et qu'il y a des dépôts limités de germoplasme de chanvre dans les banques de semences internationales. Au total, 14 031 SNP ont été identifiés après application de filtres de qualité et d'absence. Analyse en composantes principales (ACP) de la marijuana et du chanvre ( Fig 1a) a révélé une structure génétique claire séparant la marijuana et le chanvre le long du premier composant principal (PC1). Cette distinction a été soutenue en utilisant l'algorithme fastSTRUCTURE [ 6 ] en supposant K = 2 populations ancestrales ( Fig 1c ). PCA et fastSTRUCTURE ont produit des résultats très similaires: la position d'un échantillon le long de PC1 était fortement corrélée avec son appartenance à un groupe selon fastSTRUCTURE à K = 2 (r 2 = 0,98; p-value <1 x 10 -15 ).
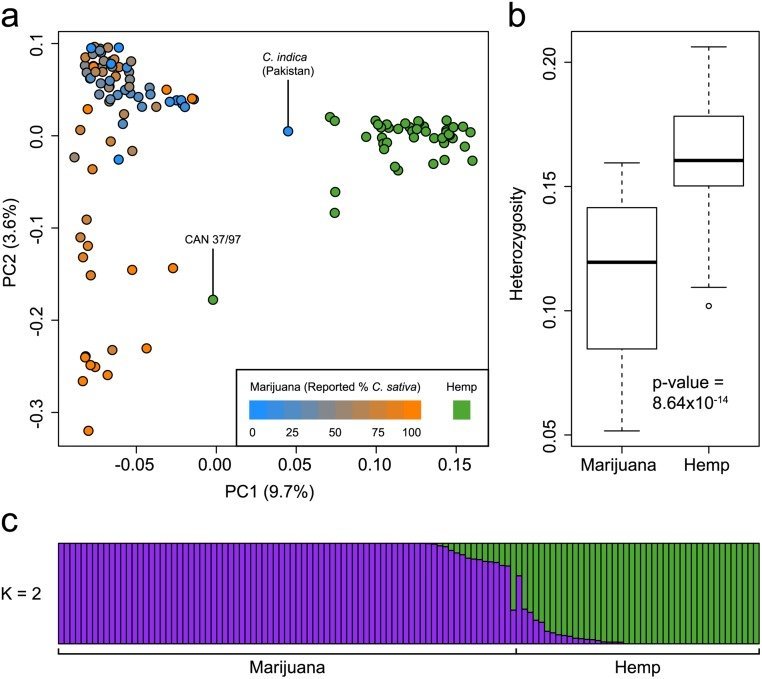
(A) PCA de 43 chanvre et 81 échantillons de marijuana en utilisant 14 031 SNP. Les échantillons de chanvre sont colorés en vert et les échantillons de marijuana sont colorés en fonction de leur C signalé . l' ascendance sativa . La proportion de la variance expliquée par chaque PC est indiquée entre parenthèses le long de chaque axe. Les deux échantillons étiquetés avec leurs identifiants sont discutés dans le texte. (b) Boxplots montrant une hétérozygosité significativement plus faible dans la marijuana que dans le chanvre. (c) Structure de la population de chanvre et de marijuana estimée en utilisant le modèle fastSTRUCTURE admixture à K = 2. Chaque échantillon est représenté par une fine ligne verticale, qui est partitionnée en deux segments colorés qui représentent l'appartenance estimée de l'échantillon dans chacun des deux groupes inférés . Les échantillons de chanvre et de marijuana sont étiquetés sous la parcelle.
On observe une putative C . indica de la marijuana du Pakistan qui est génétiquement plus proche du chanvre que des autres variétés de marijuana ( Fig 1a ). De même, l'échantillon de chanvre CAN 37/97 se rapproche davantage des souches de marijuana ( Fig 1a). Ces valeurs aberrantes peuvent être dues à la confusion dans l'échantillon ou à leur classification comme chanvre ou marijuana. L'échantillon de CAN 37/97 que nous avons génotypé provenait d'une collection canadienne de matériel génétique de chanvre, qui a obtenu cette accession de la banque de gènes IPK (Gatersleben, Allemagne). Le pays d'origine est la France, mais il y a peu d'information indiquant que le CAN 37/97 est cultivé comme chanvre. Alternativement, ces échantillons peuvent être de véritables valeurs aberrantes et représentent des souches exceptionnelles qui sont génétiquement différentes des autres dans leur groupe. En utilisant l'ensemble de données actuel, l'identification non ambiguë d'un échantillon comme étant du chanvre ou de la marijuana serait possible dans le premier cas, mais pas dans le second. En tout cas, nous trouvons que l'axe principal de la variation génétique dans le cannabis différencie le chanvre de la marijuana.
Ces résultats élargissent considérablement notre compréhension de l'évolution de la marijuana et des lignées de chanvre dans le cannabis . Des analyses antérieures ont montré que la marijuana et le chanvre diffèrent dans leur capacité de biosynthèse des cannabinoïdes, la marijuana possédant l' allèle B T codant pour l'acide tétrahydrocannabinolique synthase et le chanvre possédant typiquement l' allèle B D pour l'acide cannabidiolique synthase [ 7 ]. De même, l'analyse transcriptomique des fleurs femelles a montré que les gènes des voies cannabinoïdes sont significativement régulés positivement dans la marijuana par rapport au chanvre, comme attendu des niveaux très élevés de THC dans le premier par rapport au second [ 3].] Nos résultats indiquent que les différences génétiques entre les deux sont réparties à travers le génome et ne sont pas limitées aux locus impliqués dans la production de cannabinoïdes. En outre, nous trouvons que les niveaux d'hétérozygotie sont plus élevés dans le chanvre que dans la marijuana ( Fig 1b , Mann-Whitney U-test, p-value = 8,64 x 10 -14 ), ce qui suggère que les cultivars de chanvre proviennent d'une base génétique plus large que celle des souches de marijuana et / ou que la reproduction chez les proches parents est plus fréquente chez la marijuana que chez le chanvre.
La différence entre les plants de marijuana et de chanvre a des implications juridiques considérables dans de nombreux pays, et à ce jour, les applications médico-légales ont essentiellement consisté à déterminer si une plante devait être classée comme médicament ou non médicamenteuse [ 8 ]. Les règlements de l'UE et du Canada autorisent uniquement la culture de cultivars de chanvre contenant moins de 0,3% de THC. Alors que le chanvre et la marijuana semblent relativement bien séparés le long PC1 ( Fig 1a ), nous n'avons trouvé aucun SNP avec des différences fixes entre ces deux groupes: la valeur F ST la plus élevée entre chanvre et marijuana parmi les 14,031 SNP était de 0,87 pour un SNP avec une allèle de 0,82 dans le chanvre et 0 dans la marijuana ( tableau S1 ). Le F ST moyen entre le chanvre et la marijuana est de 0.156 ( S1 Fig), qui est similaire au degré de différenciation génétique chez les humains entre les Européens et les Asiatiques de l'Est [ 9 ]. Ainsi, alors que la culture du cannabis a entraîné une différenciation génétique claire selon l'utilisation, le chanvre et la marijuana partagent encore largement un pool commun de variations génétiques.
Bien que la séparation taxonomique des taxons putative C . sativa et C . indica reste controversée, une taxonomie vernaculaire qui distingue les souches «Sativa» et «Indica» est largement répandue dans la communauté de la marijuana. On croit généralement que les plantes de type sativa, hautes et étroites avec des feuilles étroites, produisent de la marijuana avec un effet psychoactif cérébral stimulant, tandis que les plantes de type Indica, courtes avec de larges feuilles, produiraient de la marijuana sédative et relaxante. Nous trouvons que la structure génétique de la marijuana est en accord partiel avec les estimations d'ascendance spécifiques à la souche obtenues à partir de diverses sources en ligne ( Fig 2 , S2 Table). On observe une corrélation modérée entre les positions des souches de marijuana le long de la première composante principale (PC1) de la figure 2a et les estimations déclarés de C . sativa ascendance ( figure 2c) (r 2 = 0,36; p = 2,62 x 10 -9 ). Cette relation est également observée pour la deuxième composante principale (PC2) de la figure 1a (r 2 = 0,34; p = 1,21 x 10 -8 ). Cette observation suggère que C . sativa et C. indica peut représenter des pools distincts de diversité génétique [ 1] mais cette reproduction a abouti à un mélange considérable entre les deux. Alors qu'il semble y avoir une base génétique pour l'ascendance rapportée de nombreuses souches de marijuana, dans certains cas, l'attribution de l'ascendance est fortement en désaccord avec nos données de génotype. Par exemple , nous avons constaté que la Jamaïque Agneaux Pain (100% rapporté C . Sativa ) était presque identique (IBS = 0,98) à un 100% rapporté C . Indica d'Afghanistan. La confusion des échantillons ne peut pas être exclue comme une raison potentielle de ces différences, mais un niveau similaire de classification erronée a été trouvé dans les souches obtenues dans les coffee shops néerlandais sur la base de la composition chimique [ 10].] L'inexactitude de l'ascendance rapportée dans la marijuana provient probablement de la nature essentiellement clandestine de la culture et de la culture du cannabis au cours du siècle dernier. Reconnaissant cela, les souches de marijuana vendues à des fins médicales sont souvent appelées «dominantes» Sativa ou Indica pour décrire leurs caractéristiques morphologiques et leurs effets thérapeutiques [ 10 ]. Nos résultats suggèrent que l'ascendance rapportée de certaines des variétés de marijuana les plus communes capture seulement partiellement leur véritable ascendance.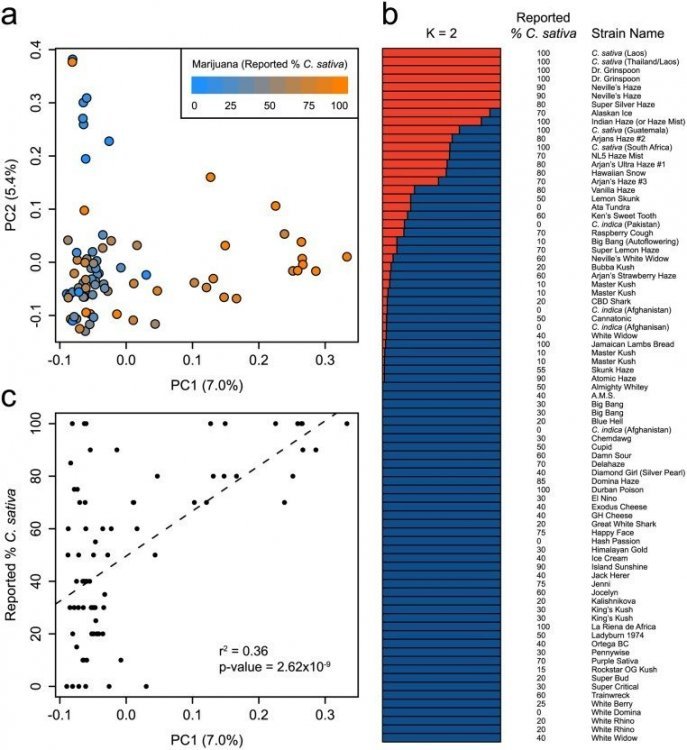
(A) PCA de 81 échantillons de marijuana en utilisant 9776 SNP. Les échantillons sont colorés en fonction de leur C signalé . l' ascendance sativa . La proportion de la variance expliquée par chaque PC est indiquée entre parenthèses le long de chaque axe. (b) Structure de la population de marijuana calculée en utilisant le modèle fastSTRUCTURE admixture à K = 2. Chaque échantillon est représenté par une barre horizontale, qui est partitionnée en deux segments colorés qui représentent l'appartenance estimée de l'échantillon dans chacun des deux groupes inférés. À côté de chaque barre est le nom et rapporté% de l'échantillon C . l' ascendance sativa . (c) La corrélation entre l'axe principal de la structure génétique (PC1) dans la marijuana et C rapporté . l'ascendance sativa .
En tant que plante dioïque pollinisée par le vent (bien qu'il existe des formes monoïques), le cannabisest très hétérozygote et de nombreuses souches de marijuana sont propagées par clonage afin de conserver leur identité génétique. Contrairement à d'autres cultures à reproduction clonale comme les pommes et les raisins, les noms de souches sont attribués aux plantes de marijuana, même si elles sont cultivées à partir de graines. Ainsi, un nom de souche de marijuana ne représente pas nécessairement une variété génétiquement unique. Pour étudier l'identité génétique de souches de marijuana nommées au niveau génétique, nous avons comparé des échantillons portant des noms identiques entre eux et avec tous les autres échantillons génotypés. Nous avons trouvé que dans 6 des 17 comparaisons (35%), les échantillons étaient plus génétiquement semblables aux échantillons portant des noms différents qu'aux échantillons portant des noms identiques. Nous concluons que l'identité génétique d'une souche de marijuana ne peut être déduite de manière fiable par son nom ou par son ascendance déclarée.
Le chanvre est toujours classé comme C . sativa dans la littérature publiée antérieurement [ 11 , 12 ], et l'hypothèse qui prévaut est que les variétés utilisées pour la production de fibres et des graines sont obtenues à partir de C . sativa [ 1 ]. Nos résultats sont incompatibles avec cette proposition: la distance génétique entre le cannabis et le chanvre est négativement corrélée avec le C rapporté . sativa ascendance (r 2 = 0,17; p-value = 0,0002) et est négativement corrélée avec sa position le long de PC1 de la figure 2a (r 2 = 0,43; p-value = 4,63 x 10 -11). De plus, nous trouvons que F ST est plus élevé entre les variétés de chanvre et de marijuana avec 100% de C rapporté . l' ascendance sativa (F ST = 0,161) qu'entre le chanvre et les souches avec 100% de C rapporté . l' ascendance indica (F ST = 0,136). Hillig (2005) a contesté la C . sativa origine de chanvre, et de noter que les souches fibre / graine de grappe Asie avec C . indica [ 1 ]. De même, une étude récente utilisant des marqueurs ADN (RAPD) Amplified Polymorphic aléatoire a constaté que C . indica plus étroitement liée au chanvre qu'avecC . sativa ou souches de marijuana hybride [ 8 ]. Conformément à ces études, nos résultats suggèrent que le chanvre a une plus grande proportion d'allèles en commun avec C . indica qu'avec C . sativa .
Il y a une pénurie de dépôts publics pour le germoplasme de chanvre et seulement un patchwork de collections privées de variétés de marijuana dans le monde entier. Ces facteurs, et la faible viabilité des semences de cannabis après un stockage prolongé, font craindre que la variation génomique que nous décrivons ici soit en danger d'être perdue. Après des décennies de réglementations restrictives et le remplacement de la fibre de chanvre par des produits synthétiques, la culture du cannabis connaît actuellement une résurgence dans de nombreuses parties du monde. Par exemple, la superficie consacrée au chanvre au Canada a atteint 27 000 ha en 2013 [ 13 ] et le soutien à la recherche sur le chanvre a été inclus dans le récent Farm Bill des États-Unis [ 14].] La présente étude fournit des éclaircissements sur la structure génétique de la marijuana et du chanvre et met en évidence les défis importants associés à la conservation du germoplasme de la marijuana en raison de son passé clandestin. L'obtention d'un système de classification pratique, précis et fiable pour le cannabis , incluant un système d'enregistrement variétal pour les plantes de type marijuana, nécessitera un investissement scientifique important et un cadre légal acceptant les formes licites et illicites de cette plante. Un tel système est essentiel pour réaliser l'énorme potentiel du cannabis en tant que culture à usages multiples (chanvre) et en tant que plante médicinale (marijuana).
Matériaux et méthodes
Matériel génétique et génotypage
Les souches de marijuana génotypées dans cette étude ont été fournies par l'auteur DH (cultivées par des producteurs autorisés de Santé Canada) et représentent du matériel génétique cultivé et utilisé pour la reproduction dans les industries de la marijuana médicale et récréative ( tableau S2 ). Les souches de chanvre ont été fournies par l'auteur JV (titulaire de permis de culture du chanvre de Santé Canada) et représentent des cultivars de semences et de fibres modernes cultivés au Canada ainsi que divers germoplasmes européens et asiatiques ( tableau S3 ). L'ADN a été extrait à partir de tissu de feuille de chanvre en utilisant un mini kit de plante Qiagen DNeasy, et à partir de feuilles de marijuana en utilisant un kit Macherey-Nagel NucleoSpin 96 Plant II avec un traitement par collecteur à vide. La préparation de la banque et le séquençage ont été réalisés en utilisant le protocole GBS publié par Sonah et al [ 15].] La séquence brute a été déposée dans l'archive de lecture de séquence NIH (SRA), sous BioProject PRJNA285813. Les SNP avec une profondeur de lecture de 10 ou plus ont été appelés en utilisant le pipeline GBS développé par Gardner et al. [ 16 ], en alignant le canSat3 C . assemblage du génome de référence sativa [ 3 ]. Un filtrage de qualité des marqueurs génétiques a été réalisé dans PLINK 1.07 [ 17 ] en éliminant les SNP avec (i) plus de 20% de manque par locus (ii) une fréquence allélique mineure inférieure à 1% et (iii) hétérozygotie excessive (équilibre de Hardy-Weinberg) p-value inférieure à 0,0001). Après filtrage, 14 031 SNP sont restés pour l'analyse.
Collecte d'ascendance de marijuana signalée
Rapportés proportions ascendance (% C . Sativa et% C . Indica ) ont été obtenus manuellement à partir des bases de données en ligne, la souche de cannabis détaillants de semences, et les producteurs sous licence de marijuana médicale ( S2 Table ). L'auteur DH a fourni des estimations d'ascendance pour 26 souches pour lesquelles aucune information en ligne n'était disponible.
Analyse de la structure de la population et de l'hétérozygotie
L'analyse en composantes principales (ACP) a été réalisée à l'aide de PLINK 1.9 ( www.cog-genomics.org/plink2 ) [ 18 ]. fastSTRUCTURE [ 6 ] a été exécuté à K = 2 et K = 3 en utilisant les paramètres par défaut pour les échantillons de chanvre et de marijuana combinés (14.031 SNP) ( Fig 1a et 1c ), et les échantillons de marijuana seuls (9776 SNP) ( Fig 2a and 2b). L'analyse à K = 2 a été réalisée pour tester la mesure dans laquelle les échantillons reflètent deux groupes distincts. D'autres valeurs de K ont été testées (non montrées), mais n'ont pas fourni d'optimisation supplémentaire ou de valeur descriptive. L'hétérozygotie par individu a été calculée dans R en divisant le nombre de sites hétérozygotes par le nombre de génotypes non-manquants pour chaque échantillon. À la suite de la méthode de génotypage utilisée dans cette étude, nous anticipons la miscallisation systématique des génotypes d'hétérozyote comme homozygotes en raison de notre seuil de profondeur de séquençage de ≥ 10 lectures (voir Fig 2 dans [ 19 ]).
Identité par l'état (IBS) analyse
La proportion d'IBS par paires entre toutes les paires d'échantillons a été calculée en utilisant PLINK 1.07. Une valeur aberrante a été exclu de cette analyse, C . indica (Pakistan), en raison de son IBS au chanvre significativement plus élevé que toutes les autres souches de marijuana (échantillon de marijuana marqué dans la figure 1a ).
Pour déterminer si la population de chanvre partagé une plus grande similitude allélique C . sativa ou C . Indica marijuana, nous avons calculé le IBS moyen par paires entre chaque souche de marijuana et toutes les souches de chanvre. Nous avons effectué cette analyse à différents seuils de fréquence allélique mineure et le résultat est resté inchangé.
-
 2
2
-
 1
1





0 Commentaire
Commentaires recommandés
Il n’y a aucun commentaire à afficher.